近日,北京大学现代农业研究院/潍坊现代农业山东省实验室/小麦育种全国重点实验室邓兴旺教授团队与南京师范大学孙林华团队合作在PNAS在线发表了题为"Single-molecule views of chromatin accessibility and structure during photomorphogenesis"的研究论文。研究基于第三代长读长测序技术,将Fiber-seq应用于植物体系,首次在单分子水平上系统解析了光信号诱导的染色质开放性动态及其表观遗传学基础。本研究在体外表达了Hia5酶,该酶可在开放的染色质纤维上引入腺嘌呤甲基化标记,结合高保真长读长测序,可在单分子分辨率下同步检测染色质可及性、核小体定位与DNA甲基化状态等。该方法克服了传统染色质开放区域测序技术分辨率低、无法解析复杂重复区域、PCR扩增存在偏好性等局限,为研究环境信号调控下的染色质动态变化提供了全新的技术路径;同时对于复杂基因组染色质可及性的研究具有重要意义,有效克服了传统二代测序在高度复杂基因组分析上的难点。

植物的光形态建成是由光信号感知、转导以及基因表达调控共同介导的复杂发育过程。光敏色素感知环境光信号后,通过关键转录因子(如PIFs和HY5等)将外界信号传递至基因组层面。然而,光在调控染色质动态变化中的具体作用机制尚未完全阐明。同时,由于植物基因组中重复序列含量高且结构复杂,基于二代测序的染色质可及性分析方法(如DNase-seq和ATAC-seq)在复杂的植物基因组中的应用受限。本研究利用Fiber-seq技术,在近核苷酸精度下解析了单分子水平光信号诱导的染色质结构变化,从而提升了植物表观基因组学研究的深度与分辨率(图1)。
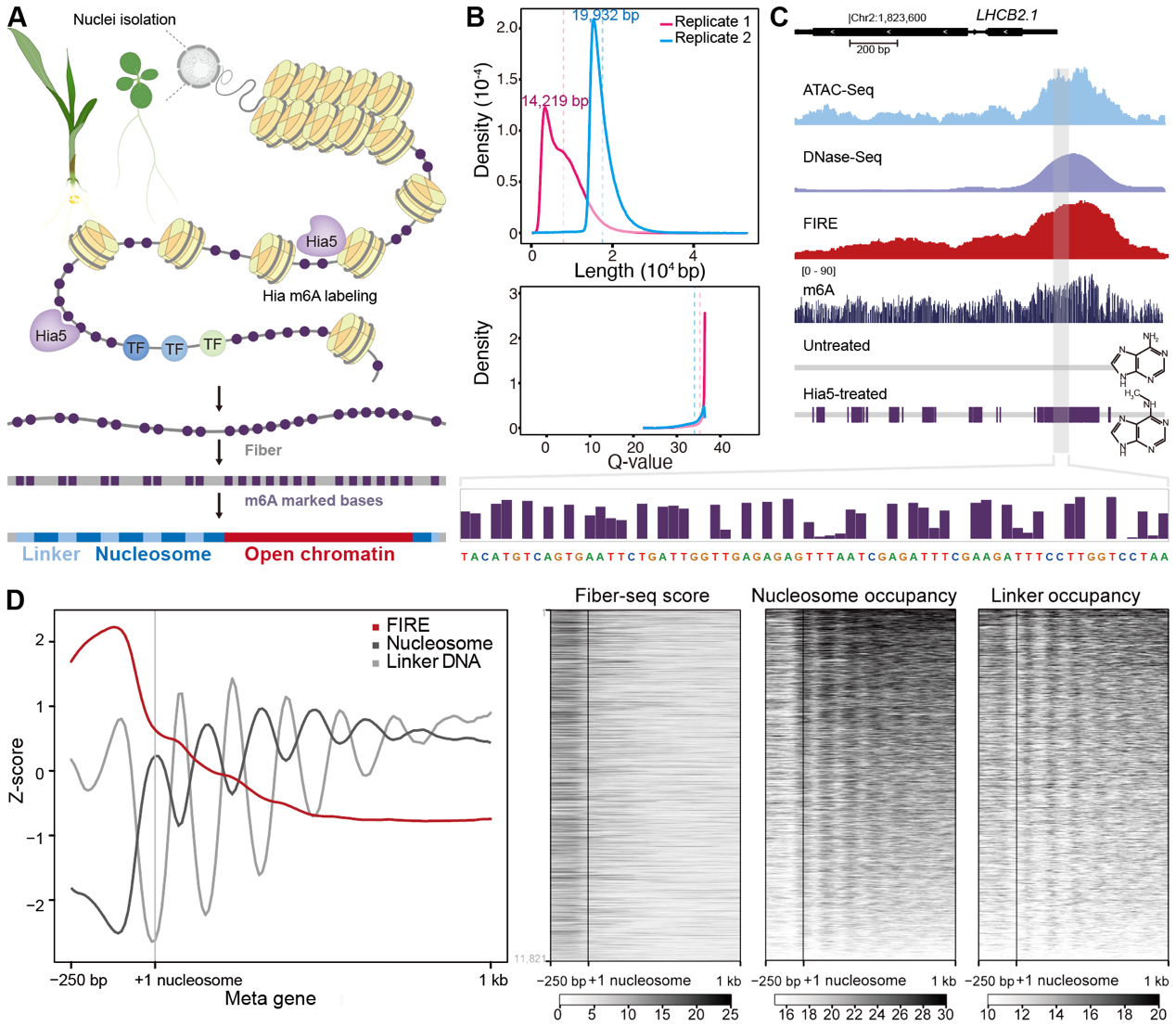
图1 Fiber-seq检测染色质可及性(A)实验流程图 (B)染色质纤维长度 (C)LHCB2.1位点的ATAC-seq、DNase-seq和Fiber-seq比较 (D)基于Fiber-seq同步检测染色质可及性和核小体定位
研究发现,在光形态建成过程中,光信号能够通过精确调控染色质的可及性与核小体重排,使染色质在特定基因组区域开放,从而影响转录调控(图2)。这些变化集中于光信号转导、激素响应、基因表达调控、转录、代谢以及生长发育等生物学功能相关的基因区域。进一步通过比较拟南芥野生型与经典光信号突变体之间的Fiber-seq数据,发现黑暗条件下的cop1-6和pifq突变体的染色质可及性与光照下的野生型类似。这验证了经典光信号通路在染色质重塑中的核心调控作用。
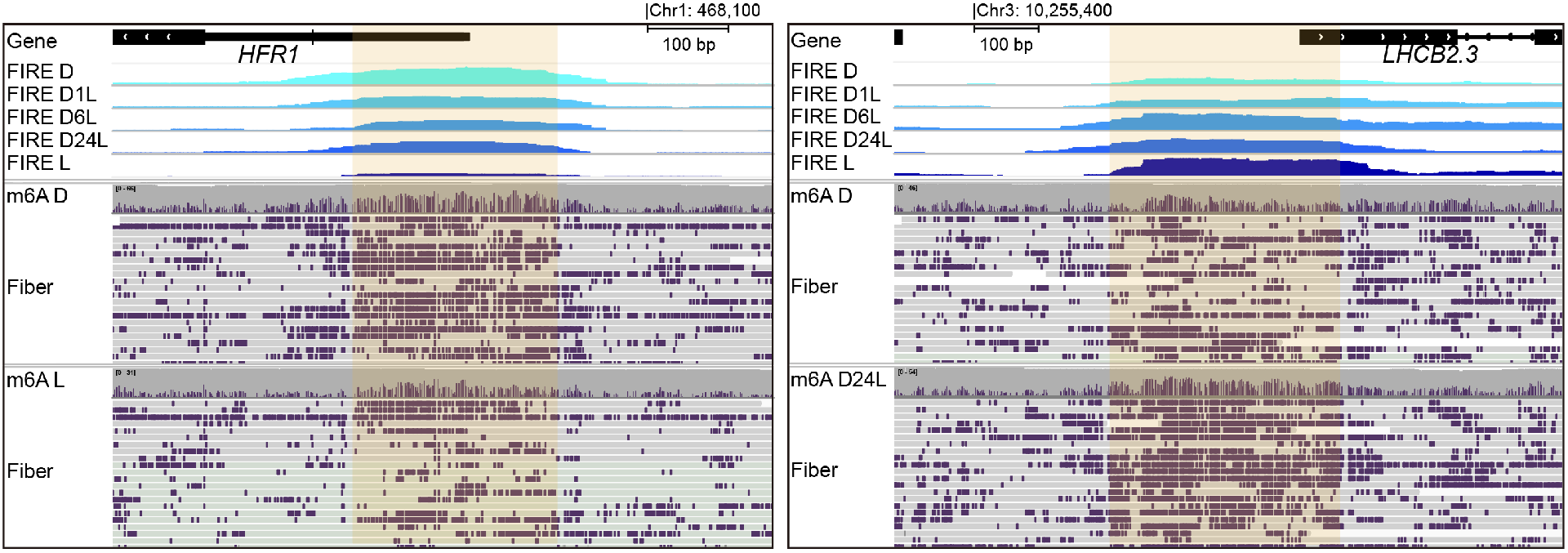
图2 转光过程中HFR1和LHCB2.3的可及性变化
此外,研究将Fiber-seq技术进一步拓展至玉米这一具有复杂基因组的植物中,展示了该方法在多倍体及高重复序列基因组中的适应性。结果表明,Fiber-seq不仅能够识别出更广泛的具有生物学意义的开放染色质区域,还能为高精度的结构变异检测与基因组从头组装提供强大的数据支撑,在T2T基因组学、乃至于超级泛基因组学时代具有重要意义(图3)。
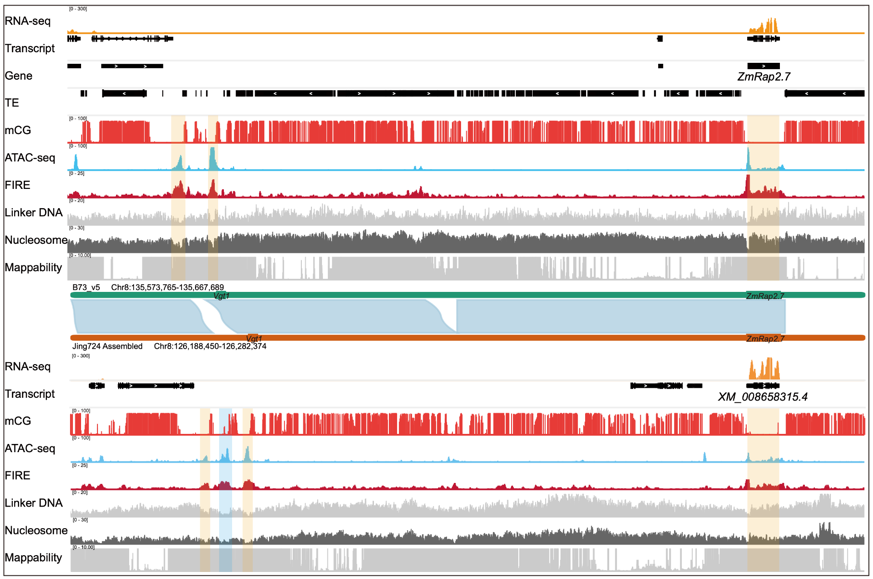
图3 利用基因组装技术检测玉米ZmRap2.7基因的结构变异,以及染色质可及性差异
综上所述,这一研究建立了适用于植物体系的Fiber-seq实验与分析平台,在技术上实现了“一个技术同步检测染色质开放性、核小体定位、DNA甲基化和基因组信息的多维整合”,为解析植物环境信号介导的表观遗传调控机制提供了新的方法学支撑,也为提升作物抗逆性与生产力的精准育种策略提供了新型工具。
北京大学现代农业研究院访问学生/南京师范大学2024级博士研究生李蕾、2024级硕士研究生陈冠宇为本文共同第一作者,科研助理张紫慧、北京大学已毕业黄铭涵博士参与了本工作,研究院何航研究员和冯铃洋研究员为本课题提供了重要帮助,邓兴旺院士、北京大学现代农业研究院邓兴旺实验室访问学者/南京师范大学孙林华研究员(曾在北京大学现代农业研究院邓兴旺实验室先后担任博士后、副研究员职位)为论文共同通讯作者。本项研究得到了山东省自然科学基金(SYS202206和ZR2022ZD22)、国家重点研发计划(2024YFA1306701)、国家自然科学基金(32230006)、山东省科技创新发展资金、江苏省高校优势学科建设工程(PAPD)的支持。特别感谢中国植物学会“青苗计划”的资助。